m6A环状RNA测序
m6A环状RNA测序近年来,科学家们发现了一种可逆的RNA甲基化--m6A,即RNA分子腺嘌呤第6位氮原子上的甲基化修饰(N6-methyladenosine,m
m6A环状RNA测序
近年来,科学家们发现了一种可逆的RNA甲基化--m6A,即RNA分子腺嘌呤第6位氮原子上的甲基化修饰(N6-methyladenosine,m6A)。研究发现,m6A是真核生物mRNA上常见的一种转录后修饰, m6A在细胞加速mRNA代谢和翻译,在细胞分化、胚胎发育和压力应答等过程中起重要作用。我们提供 m6A 甲基化免疫沉淀测序(m6A Methylation Immunoprecipitation Sequencing,简称 m6A MeRIP-seq)服务,助力 m6A 修饰研究。目前对m6A RNA流行检测手段为MeRIP-Seq技术,我们提供成熟m6A RNA甲基化MeRIP-seq服务,技术原理如下: 将甲基化RNA特异性抗体与被随机打断的RNA片段进行共孵育,抓取有甲基化修饰的片段进行测序;同时需要平行测序一个对照(Input)样本,对照样本只含有打断的RNA片段,并不添加RNA甲基化特异性抗体与其共孵育。对照样本用于消除非特异性抓取甲基化片段的背景。对比免疫共沉淀(IP)样本和对照样本(Input)中的序列片段,将RNA甲基化修饰位点定位到转录组上,并根据RNA-seq数据,计算样本中RNA甲基化程度。
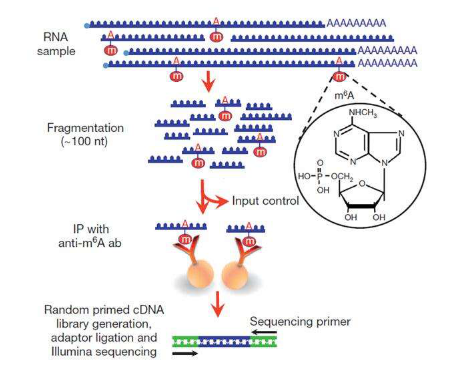
m6A RNA-seq实验流程
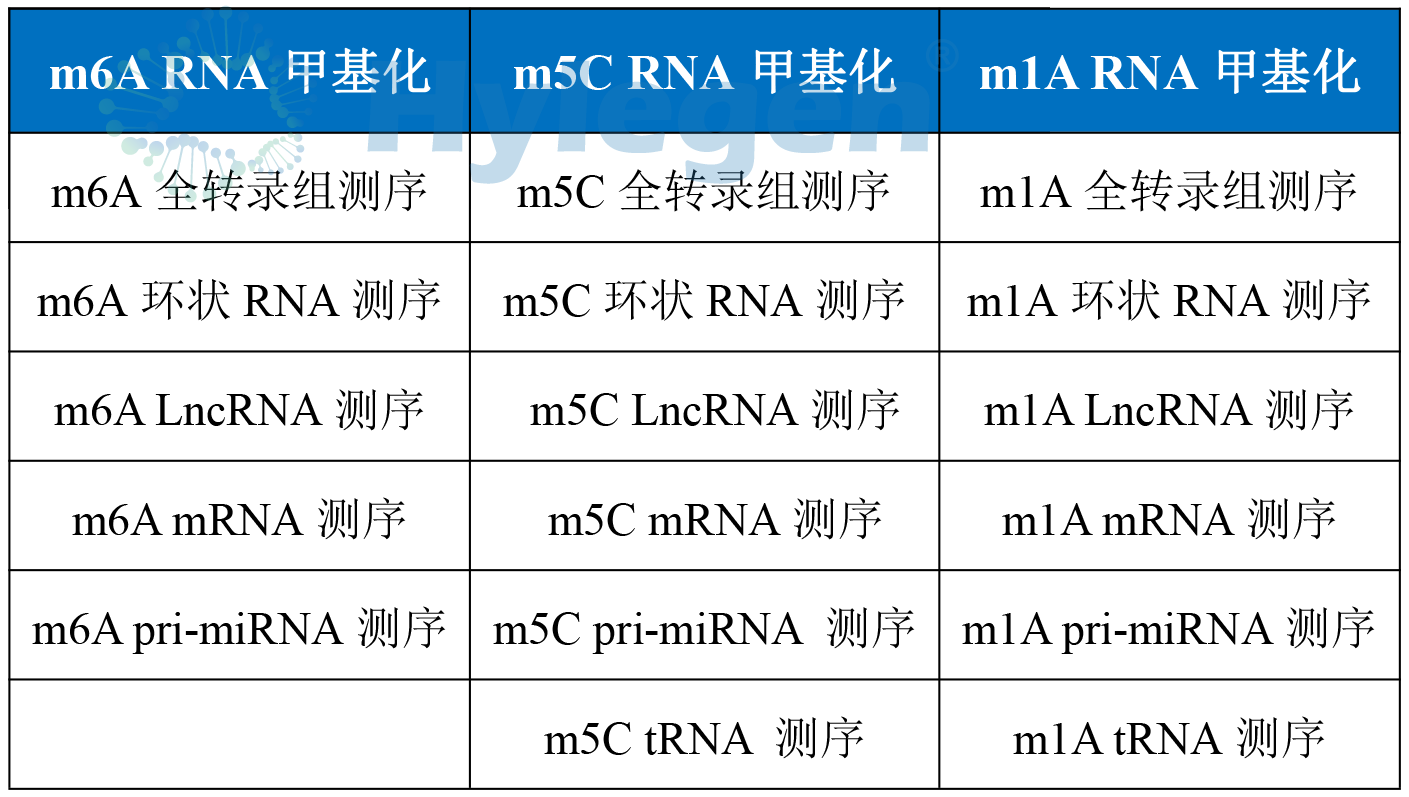
RNA甲基化测序产品
技术优势
一站式服务
客户只需提供组织、细胞、体液样品或RNA,我们为您完成从m6A RNA-seq富集,文库制备,上机测序到数据分析整套服务流程。
优化的实验流程
m6A RNA-seq的富集效率是决定数据质量的关键,我们m6A RNA-seq实验采用预验证的商业化抗体和精心优化的实验流程,具有极高的效率和特异性。
严格的质控
我们对m6A RNA-seq实验每一步骤均设有关键质控,全程监控实验质量,保证客户得到优质数据。
专业的生物信息学分析
我们拥有专业的生物信息分析团队,帮助客户进行实验数据的全面挖掘。
RNA甲基化测序与转录组联合应用
可整合RNA甲基化数据和转录组数据进行生物信息学关联分析,揭示RNA甲基化对基因表达调控的影响,深入挖掘RNA甲基化的功能。
样品要求
样品类型
细胞、组织或RNA,其他样本类型请电询。
样品量
a) 细胞:2×107
b) 组织:100 mg-1 g
c) RNA:30-300 μg(OD260/280:1.6-2.3;RNA无明显降解28S:18S>1.5或RIN>7)
样品运输与保存
样品运输:样品置于1.5mL离心管中,封口膜封好,干冰运输。
样品保存:细胞样品或新鲜组织块可用TRIZOL或RNA保护剂处理,液氮冻存后-80℃保存;RNA样品可溶于乙醇或RNA-free的超纯水中,-80℃保存,避免反复冻融。
数据分析(下列有※表示个性化分析)
1、识别甲基化富集峰
通过高通量测序和生物信息分析,识别(p <10-5)甲基化富集的基因组区域。
注:每个样品组做一个Input,以去除基因组背景,降低假阳性率
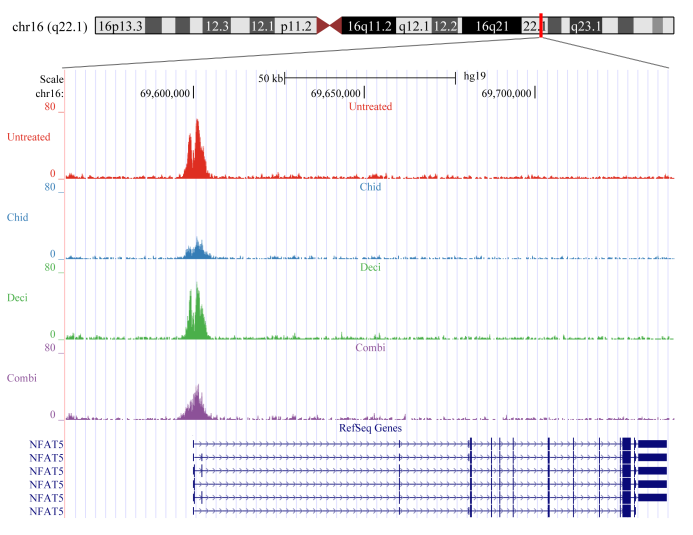
2、RNA甲基富集峰注释※
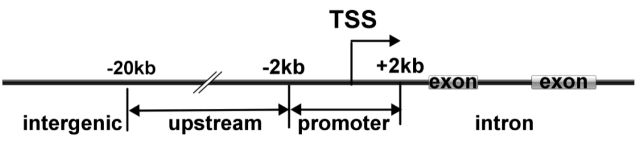
通过生物信息分析利用邻近基因对富集峰进行注释,并根据峰中点相对于已知基因的位置,将富集峰为启动子峰、上游峰、内含子峰、外显子峰、基因间峰。
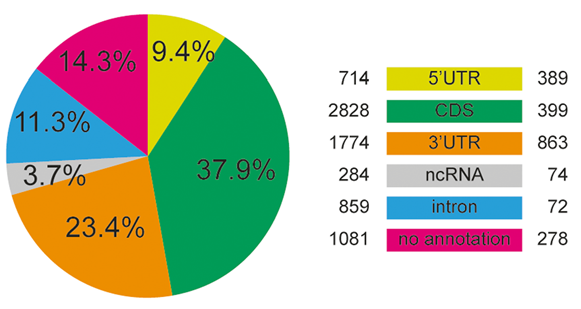
3、RNA甲基化富集峰区域的在基因组中的分布 ※
根据注释信息,绘制富集峰在不同基因组特征上的比例图。
4、RNA甲基化位点Motif分析※
RNA上甲基化的位点,可能包含某种序列motif。RNA甲基化酶可能正是通过识别这些 motif特异性进行甲基化修饰,从而完成转录后调控功能。我们成功识别了全基因组的RNA上的甲基化位点后,获取序列就能使用生物信息学的手段来搜索这些 motif,从而揭示RNA甲基化修饰的机制。

5、差异RNA甲基化区域的识别与聚类※
我们使用 diffReps 包进行差异甲基化位点的识别。默认筛选标准为 FDR<=0.05,具体以报告为准。识别样本间的差异甲基化区,发现与特定表型或疾病相关的甲基化区域。
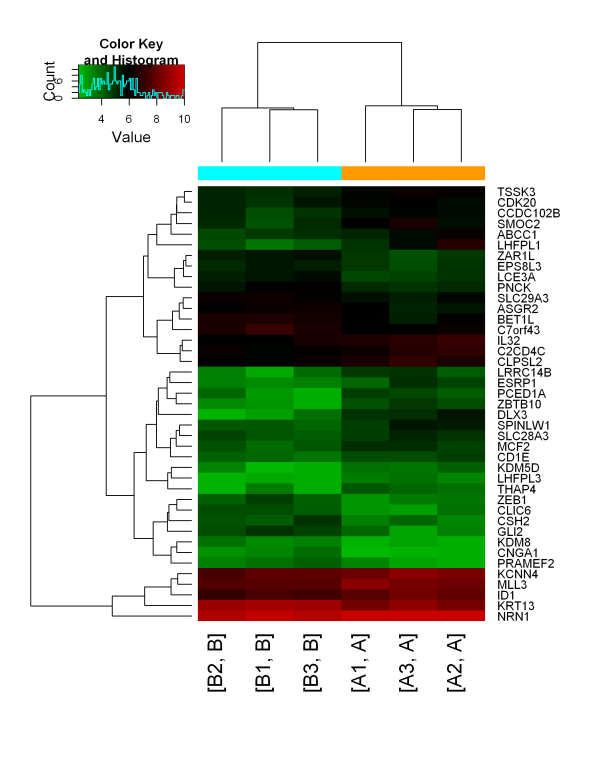
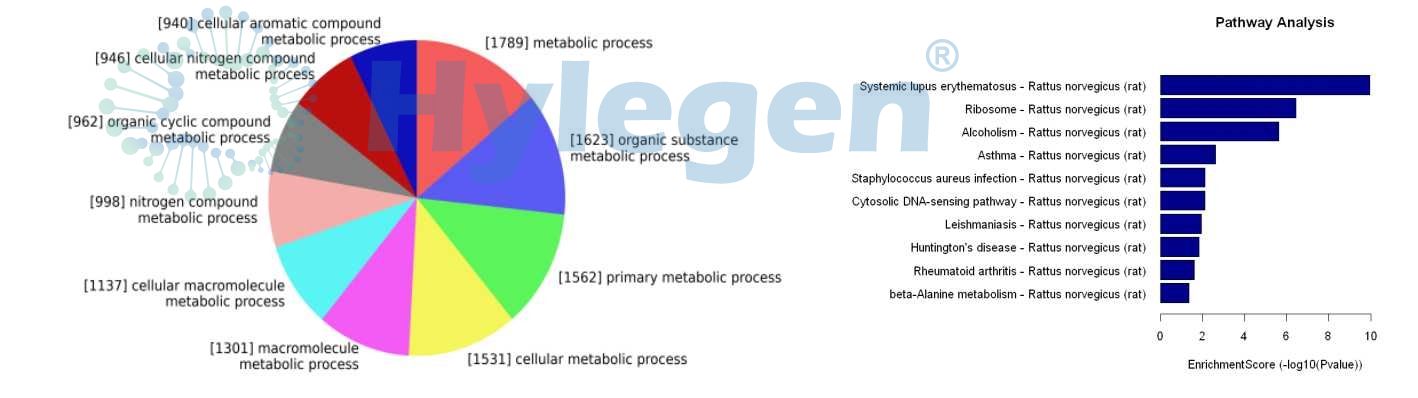
6、差异甲基化区的GO与信号通路分析
对差异甲基化基因进行功能分类,并发现明显富集的功能条目。
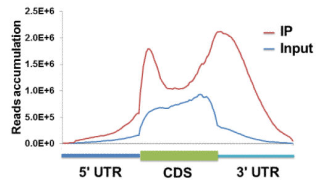
7、Reads 的分布※
使用 ngs.plot 工具可以展示匹配 reads 在 TSS, genebody, TES 等位点的分布情况,可以用来观察 RNA 甲基化对 TSS 等位点是否有偏好。
案例解析
案例1:m6A调控环状RNA编码蛋白质
原文:Extensive translation of circular RNAs driven by N6-methyladenosine
期刊:Cell Research 影响因子:15.60
这篇文章出自中科院王泽峰团队,证实发生大量的环形RNA可作为mRNA来编码蛋白,这些环形mRNA通过m6A RNA甲基化修饰驱动非帽依赖性的翻译机制来参与蛋白编码过程。该研究进一步拓展了环装RNA的功能,对蛋白质的来源的多样性有新的认识,具有十分重要的理论意义。作者借助RIP测序技术,发现与mRNA相比,m6A甲基化修饰在环状RNA上更普遍。
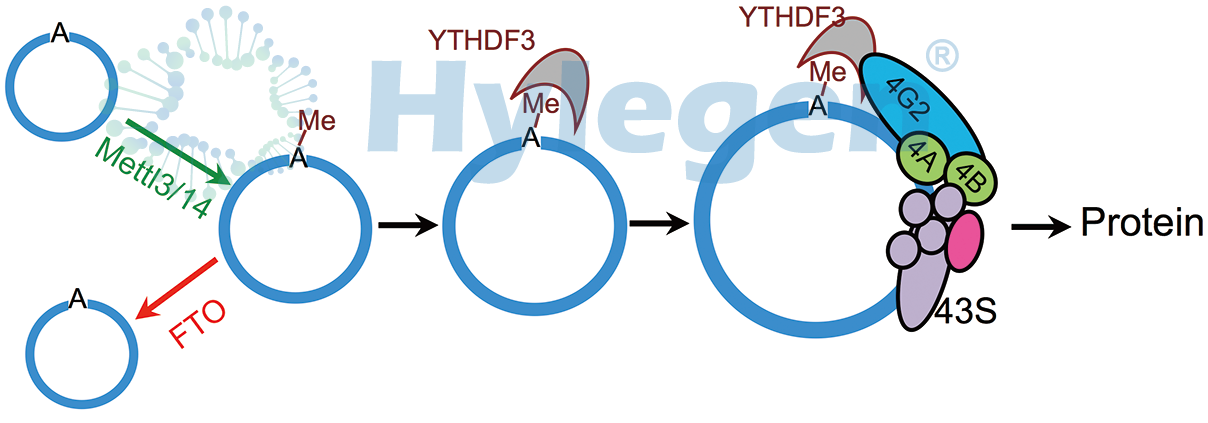
图1. m6A调控circRNA编码蛋白质机制
案例2:环状RNA普遍存在m6A修饰且呈现细胞特异性
原文:Genome-Wide Maps of m6A circRNAs Identify Widespread and Cell-Type-Specific Methylation Patterns that Are Distinct from mRNAs
期刊:Cell Reports 影响因子:8.03
本文通过高通量测序方法检测到Hela细胞和hESC细胞m6A RNA甲基化谱,发现环状RNA在两类细胞中普遍存在。通过生信分析,对比两种细胞共有及特有的环状RNA并通过验证手段(Sanger测序)验证成环状位置的序列。
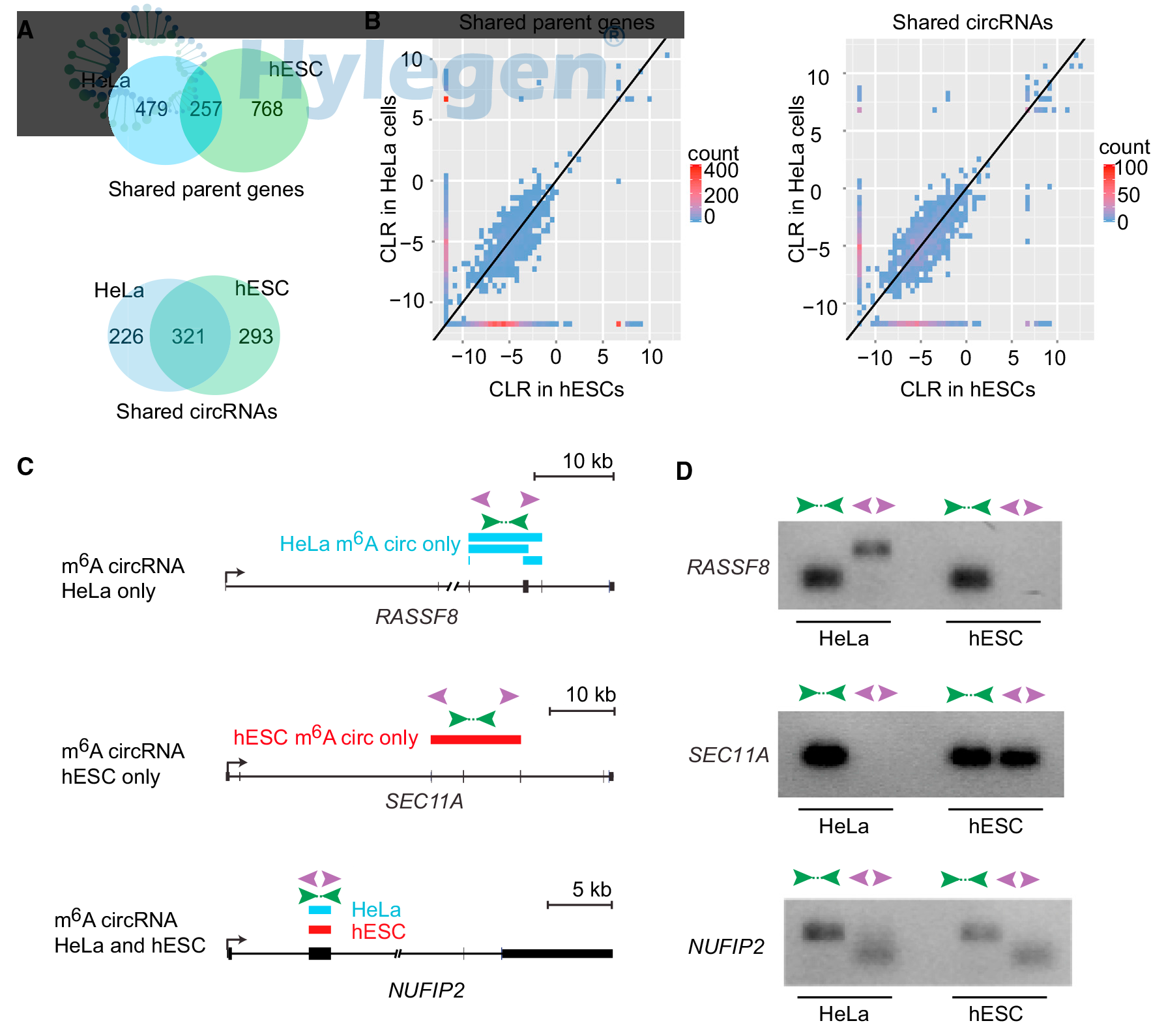
图2. Hela细胞和hESC细胞m6A甲基化谱及序列的验证